# Usando read.csv()
dados_read_csv <- read.csv("dados.csv")
# Usando read_csv() do pacote readr
library(readr)
dados_readr <- read_csv("dados.csv")3 Manipulação de dados
Dominar técnicas de manipulação e processamento de dados em R é fundamental para qualquer pessoa que trabalhe com análise de dados ou ciência de dados. A capacidade de limpar, transformar e preparar dados de maneira eficiente é essencial para garantir que os resultados das análises sejam precisos e confiáveis. Além disso, o domínio dessas técnicas economiza tempo e aumenta a produtividade.
3.1 Importar arquivos externos
Dois dos formatos mais comuns para armazenamento de dados não tão grandes são csv e xlsx.
Ao carregar dados de arquivos csv em R, duas opções comumente utilizadas são as funções read.csv() e read_csv(). Ambas são eficazes para importar dados tabulares, mas apresentam diferenças significativas. A função read.csv() é uma opção padrão no R base, sendo simples de usar e amplamente conhecida. Por outro lado, read_csv() faz parte do pacote readr, oferecendo desempenho otimizado e detecção automática de tipos de dados. Enquanto read.csv() tende a ser mais lenta, especialmente com grandes conjuntos de dados, read_csv() é mais rápida e precisa, sendo capaz de manter os nomes das colunas como símbolos e converter adequadamente os dados, inclusive lidando com strings vazias.
Para importar dados de um arquivo Excel (formato xlsx) em R, podemos usar a biblioteca readxl. Primeiro, é necessário instalá-la usando o comando install.packages("readxl"). Em seguida, podemos usar a função read_excel() para ler os dados. Por exemplo:
library(readxl)
dados <- read_excel("arquivo.xlsx")É uma boa prática definir um diretório de trabalho em seus scripts R porque isso ajuda a manter a organização e facilita o acesso aos arquivos de dados e resultados. Ao definir um diretório de trabalho, você garante que todos os arquivos referenciados em seus scripts serão encontrados facilmente, sem a necessidade de especificar caminhos absolutos longos.
Para definir o diretório de trabalho no R, você pode usar a função setwd(). Por exemplo, se você deseja definir o diretório como “C:/MeuDiretorio”, você pode fazer o seguinte:
setwd("C:/MeuDiretorio")Você pode definir o diretório usando a interface do RStudio. Basta selecionar no menu “Session” a opção “Set Working Directory” e em seguida “Choose Directory”. Isso abrirá uma caixa de diálogo onde você pode navegar até o diretório desejado e selecioná-lo. Depois de selecionar o diretório, ele se tornará o diretório de trabalho atual.
3.2 O pacote tidyverse
O pacote tidyverse é uma coleção de pacotes do R projetados para trabalhar de forma integrada e intuitiva na análise de dados. Ele inclui uma variedade de pacotes poderosos e populares, como ggplot2, dplyr, tidyr, tibble, readr, purrr, forcats e stringr. Cada pacote no tidyverse foi projetado para lidar com uma etapa específica do fluxo de trabalho de análise de dados, desde a importação e limpeza até a visualização e modelagem. Todos os pacotes no tidyverse compartilham uma filosofia de design subjacente, gramática e estruturas de dados, veja mais na página do pacote.
3.3 O operador pipe %>%
O operador %\>%, conhecido como pipe, é uma ferramenta poderosa em R que facilita a encadeamento de operações em sequência. Ele permite escrever código de forma mais clara e concisa, especialmente ao trabalhar com pacotes do tidyverse. O pipe recebe o resultado de uma expressão à esquerda e o passa como primeiro argumento para a próxima expressão à direita.
Você não precisa digitar %\>% toda vez que precisar. Utilize o atalho Ctrl+Shitf+M.
Vamos supor que temos uma função f, uma função g e uma variável x. Queremos aplicar g a x e, em seguida, aplicar f ao resultado. Aqui está como poderíamos fazer isso de duas maneiras: usando a abordagem encadeada tradicional e usando o pipe %>%.
resultado <- f(g(x))
x %>%
g() %>%
f()Ambos os métodos produzirão o mesmo resultado. No entanto, a segunda abordagem usando o pipe %>% é mais legível e fácil de entender, especialmente quando estamos encadeando múltiplas operações. Isso torna o código mais conciso e mais próximo de uma leitura natural da operação que está sendo realizada.
Uma boa prática ao usar o pipe %>% é quebrar a linha após cada pipe para melhorar a legibilidade do código.
3.4 Dados no formato tidy
“Tidy datasets are all alike, but every messy dataset is messy in its own way.” — Hadley Wickham.
Um mesmo conjunto de dados pode ser representado de diversas maneiras. Veja o código abaixo que mostra o mesmo dado em três diferentes formatos.
table1# A tibble: 6 × 4
country year cases population
<chr> <dbl> <dbl> <dbl>
1 Afghanistan 1999 745 19987071
2 Afghanistan 2000 2666 20595360
3 Brazil 1999 37737 172006362
4 Brazil 2000 80488 174504898
5 China 1999 212258 1272915272
6 China 2000 213766 1280428583table2# A tibble: 12 × 4
country year type count
<chr> <dbl> <chr> <dbl>
1 Afghanistan 1999 cases 745
2 Afghanistan 1999 population 19987071
3 Afghanistan 2000 cases 2666
4 Afghanistan 2000 population 20595360
5 Brazil 1999 cases 37737
6 Brazil 1999 population 172006362
7 Brazil 2000 cases 80488
8 Brazil 2000 population 174504898
9 China 1999 cases 212258
10 China 1999 population 1272915272
11 China 2000 cases 213766
12 China 2000 population 1280428583table4a# A tibble: 3 × 3
country `1999` `2000`
<chr> <dbl> <dbl>
1 Afghanistan 745 2666
2 Brazil 37737 80488
3 China 212258 213766Todas as representações acima são dos mesmos dados, mas não são igualmente fáceis de utilizar. A table1, por exemplo, será muito mais acessível para trabalhar dentro do tidyverse devido à sua organização no formato tidy. Existem três regras inter-relacionadas que caracterizam um conjunto de dados no formato tidy:
- Cada variável é uma coluna; cada coluna representa uma variável.
- Cada observação é uma linha; cada linha representa uma observação.
- Cada valor é uma célula; cada célula contém um único valor.
A figura abaixo representa graficamente este conceito.

A pivotação de dados é o processo de reorganizar um conjunto de dados para torná-lo compatível com o formato tidy. Isso envolve transformar os dados de um formato mais largo para um formato mais longo, ou vice-versa, para garantir que cada variável corresponda a uma coluna e cada observação a uma linha.
No exemplo abaixo, estamos transformando os dados de table2 para um formato mais largo, onde cada valor único da variável type se torna uma nova coluna. Note que cada unidade de informação (país, ano, casos e contagem) está quebrado em duas linhas. Então essa operação deixa a tabela de dados mais larga, garantindo que cada unidade de dado esteja representada em uma única linha.
table2 %>%
pivot_wider(names_from="type", values_from="count")# A tibble: 6 × 4
country year cases population
<chr> <dbl> <dbl> <dbl>
1 Afghanistan 1999 745 19987071
2 Afghanistan 2000 2666 20595360
3 Brazil 1999 37737 172006362
4 Brazil 2000 80488 174504898
5 China 1999 212258 1272915272
6 China 2000 213766 1280428583No exemplo abaixo, estamos transformando os dados de table4a em um formato mais longo, onde as colunas representando anos específicos (1999 e 2000) são reunidas em uma única coluna chamada year, e os valores correspondentes são colocados em uma nova coluna chamada cases. Neste caso, a informação sobre os anos estavam armazenadas como nome de colunas, mas, pelo princípio de dados tidy, deveriam estar em colunas. Por isso, utilizamos a função pivot_longer.
table4a %>%
pivot_longer(cols = c(`1999`, `2000`), names_to = "year", values_to = "cases")# A tibble: 6 × 3
country year cases
<chr> <chr> <dbl>
1 Afghanistan 1999 745
2 Afghanistan 2000 2666
3 Brazil 1999 37737
4 Brazil 2000 80488
5 China 1999 212258
6 China 2000 213766As duas funções pivot_wider e pivot_longer são suficientes para fazer a transformação de bases de dados no formato tidy.
3.5 Principais verbos do pacote dplyr
O pacote dplyr é uma das ferramentas mais poderosas para manipulação de dados no ambiente R. Ele oferece um conjunto coeso de funções que simplificam tarefas comuns de manipulação, como filtragem, seleção, agrupamento, ordenação e resumo de dados. O dplyr utiliza uma sintaxe intuitiva e consistente, facilitando a escrita de código limpo e legível.
A seguir, vamos estudar o funcionamento dos principais verbos do pacote. Para exemplificar, vamos utilizar a base de dados gapminder. Ela é uma coleção de informações socioeconômicas de diversos países ao longo do tempo, veja Rosling (2012). Ela inclui variáveis como expectativa de vida, PIB per capita, taxa de mortalidade infantil e tamanho da população para diferentes países e anos, cobrindo um período de várias décadas.
Para carregar a base de dados gapminder, você precisa carregar o pacote gapminder. Com o pacote gapminder carregado, a base de dados gapminder estará disponível para uso em seu ambiente R:
library(gapminder)Warning: package 'gapminder' was built under R version 4.2.3head(gapminder)A função glimpse() fornece uma visão geral rápida e concisa da estrutura de um conjunto de dados. Quando aplicada a um conjunto de dados, como o gapminder, ela exibe informações essenciais sobre as variáveis presentes, incluindo a quantidade de linhas, colunas e as primeiras linhas do conjunto de dados:
glimpse(gapminder)Rows: 1,704
Columns: 6
$ country <fct> "Afghanistan", "Afghanistan", "Afghanistan", "Afghanistan", …
$ continent <fct> Asia, Asia, Asia, Asia, Asia, Asia, Asia, Asia, Asia, Asia, …
$ year <int> 1952, 1957, 1962, 1967, 1972, 1977, 1982, 1987, 1992, 1997, …
$ lifeExp <dbl> 28.801, 30.332, 31.997, 34.020, 36.088, 38.438, 39.854, 40.8…
$ pop <int> 8425333, 9240934, 10267083, 11537966, 13079460, 14880372, 12…
$ gdpPercap <dbl> 779.4453, 820.8530, 853.1007, 836.1971, 739.9811, 786.1134, …3.5.1 select
O verbo select() é utilizado para selecionar colunas específicas de um conjunto de dados. Com o select(), é possível escolher as colunas desejadas com base em seus nomes, tipos de dados ou outros critérios.
Por exemplo, considerando a base de dados gapminder, suponha que desejamos selecionar apenas as colunas referentes ao ano, ao país, à expectativa de vida e ao PIB per capita. Podemos fazer isso da seguinte maneira:
# Selecionando as colunas pelo nome
gapminder %>%
select(year, country, lifeExp, gdpPercap)
# Selecionando apenas as colunas numéricas
gapminder %>%
select(where(is.numeric))
# Selecionando colunas que começam com "co"
gapminder %>%
select(starts_with("co"))Note que nos exemplos acima, nenhuma das seleções foi salva em variável. Para salvar as seleções em uma variável, você pode atribuir o resultado de cada operação select() a uma variável separada. Por exemplo:
gapminder_character <- gapminder %>%
select(where(is.character))3.5.2 arrange
O verbo arrange() é usado para reorganizar as linhas de um conjunto de dados com base nos valores de uma ou mais colunas. Quando aplicado a um conjunto de dados, o arrange() classifica as linhas em ordem crescente ou decrescente com base nos valores das colunas especificadas.
No primeiro exemplo usando o verbo select(), podemos ordenar os dados por país em ordem alfabética, podemos fazer assim:
gapminder %>%
select(year, country, lifeExp, gdpPercap, pop) %>%
arrange(country)# A tibble: 1,704 × 5
year country lifeExp gdpPercap pop
<int> <fct> <dbl> <dbl> <int>
1 1952 Afghanistan 28.8 779. 8425333
2 1957 Afghanistan 30.3 821. 9240934
3 1962 Afghanistan 32.0 853. 10267083
4 1967 Afghanistan 34.0 836. 11537966
5 1972 Afghanistan 36.1 740. 13079460
6 1977 Afghanistan 38.4 786. 14880372
7 1982 Afghanistan 39.9 978. 12881816
8 1987 Afghanistan 40.8 852. 13867957
9 1992 Afghanistan 41.7 649. 16317921
10 1997 Afghanistan 41.8 635. 22227415
# ℹ 1,694 more rowsNo exemplo abaixo, estamos organizando de acordo com o ano em ordem crescente e a expectativa de vida em ordem decrescente dentro de cada ano.
gapminder %>%
select(year, country, lifeExp, gdpPercap, pop) %>%
arrange(year, desc(lifeExp))# A tibble: 1,704 × 5
year country lifeExp gdpPercap pop
<int> <fct> <dbl> <dbl> <int>
1 1952 Norway 72.7 10095. 3327728
2 1952 Iceland 72.5 7268. 147962
3 1952 Netherlands 72.1 8942. 10381988
4 1952 Sweden 71.9 8528. 7124673
5 1952 Denmark 70.8 9692. 4334000
6 1952 Switzerland 69.6 14734. 4815000
7 1952 New Zealand 69.4 10557. 1994794
8 1952 United Kingdom 69.2 9980. 50430000
9 1952 Australia 69.1 10040. 8691212
10 1952 Canada 68.8 11367. 14785584
# ℹ 1,694 more rowsAo utilizar o verbo select() com o prefixo -, você pode especificar as colunas que deseja excluir do conjunto de dados. No exemplo abaixo, vamos excluir a coluna continent da seleção no conjunto de dados.
gapminder %>%
select(-continent)# A tibble: 1,704 × 5
country year lifeExp pop gdpPercap
<fct> <int> <dbl> <int> <dbl>
1 Afghanistan 1952 28.8 8425333 779.
2 Afghanistan 1957 30.3 9240934 821.
3 Afghanistan 1962 32.0 10267083 853.
4 Afghanistan 1967 34.0 11537966 836.
5 Afghanistan 1972 36.1 13079460 740.
6 Afghanistan 1977 38.4 14880372 786.
7 Afghanistan 1982 39.9 12881816 978.
8 Afghanistan 1987 40.8 13867957 852.
9 Afghanistan 1992 41.7 16317921 649.
10 Afghanistan 1997 41.8 22227415 635.
# ℹ 1,694 more rows3.5.3 filter
Para analisar dados específicos de interesse, muitas vezes é necessário filtrar o conjunto de dados para incluir apenas as observações relevantes. O verbo filter() é usado para fazer isso. Basta definir uma ou mais condições lógicas que as linhas da base de dados devem satisfazer para serem mostradas.
No exemplo abaixo, estamos filtrando os dados para incluir apenas as observações onde o país é “Brasil” ou “Argentina”.
gapminder %>%
select(year, country, lifeExp, gdpPercap, pop) %>%
arrange(year, desc(lifeExp)) %>%
filter(country == "Brazil" | country == "Argentina")# A tibble: 24 × 5
year country lifeExp gdpPercap pop
<int> <fct> <dbl> <dbl> <int>
1 1952 Argentina 62.5 5911. 17876956
2 1952 Brazil 50.9 2109. 56602560
3 1957 Argentina 64.4 6857. 19610538
4 1957 Brazil 53.3 2487. 65551171
5 1962 Argentina 65.1 7133. 21283783
6 1962 Brazil 55.7 3337. 76039390
7 1967 Argentina 65.6 8053. 22934225
8 1967 Brazil 57.6 3430. 88049823
9 1972 Argentina 67.1 9443. 24779799
10 1972 Brazil 59.5 4986. 100840058
# ℹ 14 more rows3.5.4 mutate
O verbo mutate() é usado para criar ou modificar colunas em um conjunto de dados existente. Ele permite adicionar novas variáveis calculadas com base em variáveis existentes ou modificar as variáveis existentes de acordo com alguma lógica específica.
Por exemplo, podemos usar o mutate() para calcular uma nova variável que represente o PIB total de cada país multiplicando o PIB per capita pelo tamanho da população. Aqui está um exemplo de como fazer isso com o conjunto de dados gapminder:
gapminder_total_gdp <- gapminder %>%
select(country, year, lifeExp, gdpPercap, pop) %>%
mutate(total_gdp = gdpPercap * pop)3.5.5 summarise
O verbo summarise() é usado para resumir os dados em uma única linha, geralmente calculando estatísticas resumidas como média, soma, mediana, etc. Ele permite calcular resumos estatísticos em um conjunto de dados, criando uma nova tabela contendo os resultados resumidos.
Aqui está um exemplo de como usar summarise() para calcular a média da expectativa de vida usando os dados do gapminder:
gapminder %>%
summarise(mean_lifeExp = mean(lifeExp, na.rm = TRUE))# A tibble: 1 × 1
mean_lifeExp
<dbl>
1 59.53.5.6 group by
O verbo group_by() é usado para dividir os dados em grupos com base nos valores de uma ou mais variáveis. Ele não realiza cálculos por si só, mas altera o comportamento das funções de resumo, como summarise(), para operar em cada grupo separadamente.
Aqui está um exemplo de como usar group_by() com os dados do gapminder para calcular a média da expectativa de vida por continente:
gapminder %>%
group_by(continent) %>%
summarise(mean_lifeExp = mean(lifeExp, na.rm = TRUE))# A tibble: 5 × 2
continent mean_lifeExp
<fct> <dbl>
1 Africa 48.9
2 Americas 64.7
3 Asia 60.1
4 Europe 71.9
5 Oceania 74.3O exemplo abaixo utiliza todos os principais verbos do dplyr para calcular a expectativa de vida média e o PIB (em milhares) médio por continente no ano de 2007.
gapminder %>%
select(country, continent, year, lifeExp, gdpPercap) %>%
filter(year == 2007) %>% # apenas os dados para o ano de 2007
mutate(gdp = gdpPercap / 1000) %>% # representa o PIB per capita em milhares
group_by(continent) %>% # agrupar os dados por continente
summarise(mean_lifeExp = mean(lifeExp, na.rm = TRUE), # média da expectativa de vida
mean_gdp = mean(gdp, na.rm = TRUE)) %>% #média do PIB per capita em bilhões
arrange(desc(mean_lifeExp))# A tibble: 5 × 3
continent mean_lifeExp mean_gdp
<fct> <dbl> <dbl>
1 Oceania 80.7 29.8
2 Europe 77.6 25.1
3 Americas 73.6 11.0
4 Asia 70.7 12.5
5 Africa 54.8 3.09O gráfico abaixo mostra a evolução da expectativa de vida média nos continentes ao longo dos anos.
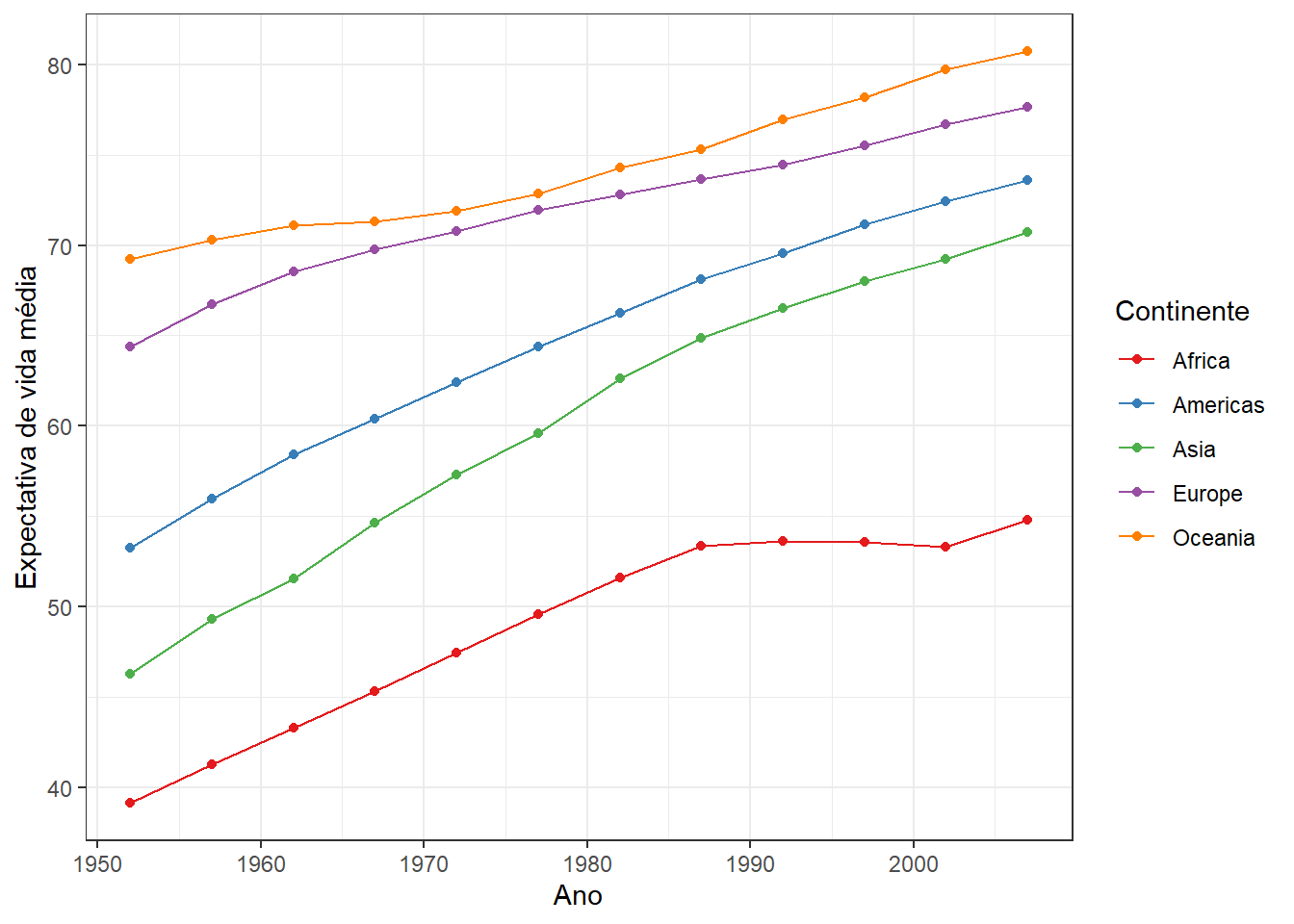
Qual mudança foi feita no código do exemplo anterior para construir os dados usados na geração deste gráfico?
3.6 Funções auxiliares
Apresentando funções auxiliares do pacote dplyr que podem ser muito úteis em diversos contextos.
pull,distinct,unite,separate_wider_delim, e a familia de funçõesslice_*.
gapminder %>%
filter(year == 1952) %>%
pull(continent) [1] Asia Europe Africa Africa Americas Oceania Europe Asia
[9] Asia Europe Africa Americas Europe Africa Americas Europe
[17] Africa Africa Asia Africa Americas Africa Africa Americas
[25] Asia Americas Africa Africa Africa Americas Africa Europe
[33] Americas Europe Europe Africa Americas Americas Africa Americas
[41] Africa Africa Africa Europe Europe Africa Africa Europe
[49] Africa Europe Americas Africa Africa Americas Americas Asia
[57] Europe Europe Asia Asia Asia Asia Europe Asia
[65] Europe Americas Asia Asia Africa Asia Asia Asia
[73] Asia Africa Africa Africa Africa Africa Asia Africa
[81] Africa Africa Americas Asia Europe Africa Africa Asia
[89] Africa Asia Europe Oceania Americas Africa Africa Europe
[97] Asia Asia Americas Americas Americas Asia Europe Europe
[105] Americas Africa Europe Africa Africa Asia Africa Europe
[113] Africa Asia Europe Europe Africa Africa Europe Asia
[121] Africa Africa Europe Europe Asia Asia Africa Asia
[129] Africa Americas Africa Europe Africa Europe Americas Americas
[137] Americas Asia Asia Asia Africa Africa
Levels: Africa Americas Asia Europe Oceaniagapminder %>%
distinct(continent)# A tibble: 5 × 1
continent
<fct>
1 Asia
2 Europe
3 Africa
4 Americas
5 Oceania gapminder %>%
slice(1:10)# A tibble: 10 × 6
country continent year lifeExp pop gdpPercap
<fct> <fct> <int> <dbl> <int> <dbl>
1 Afghanistan Asia 1952 28.8 8425333 779.
2 Afghanistan Asia 1957 30.3 9240934 821.
3 Afghanistan Asia 1962 32.0 10267083 853.
4 Afghanistan Asia 1967 34.0 11537966 836.
5 Afghanistan Asia 1972 36.1 13079460 740.
6 Afghanistan Asia 1977 38.4 14880372 786.
7 Afghanistan Asia 1982 39.9 12881816 978.
8 Afghanistan Asia 1987 40.8 13867957 852.
9 Afghanistan Asia 1992 41.7 16317921 649.
10 Afghanistan Asia 1997 41.8 22227415 635.gapminder %>%
slice_head(n = 5)# A tibble: 5 × 6
country continent year lifeExp pop gdpPercap
<fct> <fct> <int> <dbl> <int> <dbl>
1 Afghanistan Asia 1952 28.8 8425333 779.
2 Afghanistan Asia 1957 30.3 9240934 821.
3 Afghanistan Asia 1962 32.0 10267083 853.
4 Afghanistan Asia 1967 34.0 11537966 836.
5 Afghanistan Asia 1972 36.1 13079460 740.gapminder %>%
slice_tail(n = 5)# A tibble: 5 × 6
country continent year lifeExp pop gdpPercap
<fct> <fct> <int> <dbl> <int> <dbl>
1 Zimbabwe Africa 1987 62.4 9216418 706.
2 Zimbabwe Africa 1992 60.4 10704340 693.
3 Zimbabwe Africa 1997 46.8 11404948 792.
4 Zimbabwe Africa 2002 40.0 11926563 672.
5 Zimbabwe Africa 2007 43.5 12311143 470.set.seed(1)
gapminder %>%
slice_sample(n = 10)# A tibble: 10 × 6
country continent year lifeExp pop gdpPercap
<fct> <fct> <int> <dbl> <int> <dbl>
1 Montenegro Europe 1992 75.4 621621 7003.
2 Hungary Europe 1982 69.4 10705535 12546.
3 Benin Africa 1992 53.9 4981671 1191.
4 Malawi Africa 1977 43.8 5637246 663.
5 Thailand Asia 1992 67.3 56667095 4617.
6 El Salvador Americas 1962 52.3 2747687 3777.
7 China Asia 2002 72.0 1280400000 3119.
8 Chad Africa 1977 47.4 4388260 1134.
9 Peru Americas 2002 69.9 26769436 5909.
10 Senegal Africa 2002 61.6 10870037 1520.gapminder %>%
filter(year == 2007) %>%
slice_max(lifeExp, n = 2)# A tibble: 2 × 6
country continent year lifeExp pop gdpPercap
<fct> <fct> <int> <dbl> <int> <dbl>
1 Japan Asia 2007 82.6 127467972 31656.
2 Hong Kong, China Asia 2007 82.2 6980412 39725.gapminder %>%
filter(year == 2007) %>%
slice_min(lifeExp, n = 2)# A tibble: 2 × 6
country continent year lifeExp pop gdpPercap
<fct> <fct> <int> <dbl> <int> <dbl>
1 Swaziland Africa 2007 39.6 1133066 4513.
2 Mozambique Africa 2007 42.1 19951656 824.gapminder %>%
filter(year == 2007 | year == 1952) %>%
group_by(year) %>%
slice_max(lifeExp, n = 2)# A tibble: 4 × 6
# Groups: year [2]
country continent year lifeExp pop gdpPercap
<fct> <fct> <int> <dbl> <int> <dbl>
1 Norway Europe 1952 72.7 3327728 10095.
2 Iceland Europe 1952 72.5 147962 7268.
3 Japan Asia 2007 82.6 127467972 31656.
4 Hong Kong, China Asia 2007 82.2 6980412 39725.gapminder_united <- gapminder %>%
unite("country_continent", c(country, continent),
sep = "_",
remove = TRUE,
na.rm = FALSE)
gapminder_united %>%
separate_wider_delim(country_continent,
delim = "_",
names = c("country", "continent"))# A tibble: 1,704 × 6
country continent year lifeExp pop gdpPercap
<chr> <chr> <int> <dbl> <int> <dbl>
1 Afghanistan Asia 1952 28.8 8425333 779.
2 Afghanistan Asia 1957 30.3 9240934 821.
3 Afghanistan Asia 1962 32.0 10267083 853.
4 Afghanistan Asia 1967 34.0 11537966 836.
5 Afghanistan Asia 1972 36.1 13079460 740.
6 Afghanistan Asia 1977 38.4 14880372 786.
7 Afghanistan Asia 1982 39.9 12881816 978.
8 Afghanistan Asia 1987 40.8 13867957 852.
9 Afghanistan Asia 1992 41.7 16317921 649.
10 Afghanistan Asia 1997 41.8 22227415 635.
# ℹ 1,694 more rows3.7 Exercícios
Vamos trabalhar com o conjunto de dados billboard. Neste conjunto de dados, cada observação é uma música. As três primeiras colunas (artista, faixa e data de entrada) são variáveis que descrevem a música. Em seguida, temos 76 colunas (wk1-wk76) que descrevem o ranking da música em cada semana. Aqui, os nomes das colunas são uma variável (a semana) e os valores das células são outra (o ranking).
library(tidyverse)
billboard# A tibble: 317 × 79
artist track date.entered wk1 wk2 wk3 wk4 wk5 wk6 wk7 wk8
<chr> <chr> <date> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
1 2 Pac Baby… 2000-02-26 87 82 72 77 87 94 99 NA
2 2Ge+her The … 2000-09-02 91 87 92 NA NA NA NA NA
3 3 Doors D… Kryp… 2000-04-08 81 70 68 67 66 57 54 53
4 3 Doors D… Loser 2000-10-21 76 76 72 69 67 65 55 59
5 504 Boyz Wobb… 2000-04-15 57 34 25 17 17 31 36 49
6 98^0 Give… 2000-08-19 51 39 34 26 26 19 2 2
7 A*Teens Danc… 2000-07-08 97 97 96 95 100 NA NA NA
8 Aaliyah I Do… 2000-01-29 84 62 51 41 38 35 35 38
9 Aaliyah Try … 2000-03-18 59 53 38 28 21 18 16 14
10 Adams, Yo… Open… 2000-08-26 76 76 74 69 68 67 61 58
# ℹ 307 more rows
# ℹ 68 more variables: wk9 <dbl>, wk10 <dbl>, wk11 <dbl>, wk12 <dbl>,
# wk13 <dbl>, wk14 <dbl>, wk15 <dbl>, wk16 <dbl>, wk17 <dbl>, wk18 <dbl>,
# wk19 <dbl>, wk20 <dbl>, wk21 <dbl>, wk22 <dbl>, wk23 <dbl>, wk24 <dbl>,
# wk25 <dbl>, wk26 <dbl>, wk27 <dbl>, wk28 <dbl>, wk29 <dbl>, wk30 <dbl>,
# wk31 <dbl>, wk32 <dbl>, wk33 <dbl>, wk34 <dbl>, wk35 <dbl>, wk36 <dbl>,
# wk37 <dbl>, wk38 <dbl>, wk39 <dbl>, wk40 <dbl>, wk41 <dbl>, wk42 <dbl>, …a) Aplique uma tranformação na base de dados para deixá-la no formato abaixo.
# A tibble: 24,092 × 5
artist track date.entered week rank
<chr> <chr> <date> <chr> <dbl>
1 2 Pac Baby Don't Cry (Keep... 2000-02-26 wk1 87
2 2 Pac Baby Don't Cry (Keep... 2000-02-26 wk2 82
3 2 Pac Baby Don't Cry (Keep... 2000-02-26 wk3 72
4 2 Pac Baby Don't Cry (Keep... 2000-02-26 wk4 77
5 2 Pac Baby Don't Cry (Keep... 2000-02-26 wk5 87
6 2 Pac Baby Don't Cry (Keep... 2000-02-26 wk6 94
7 2 Pac Baby Don't Cry (Keep... 2000-02-26 wk7 99
8 2 Pac Baby Don't Cry (Keep... 2000-02-26 wk8 NA
9 2 Pac Baby Don't Cry (Keep... 2000-02-26 wk9 NA
10 2 Pac Baby Don't Cry (Keep... 2000-02-26 wk10 NA
# ℹ 24,082 more rowsb) Observe o resultado do item a). O que acontece se uma música estiver no top 100 por menos de 76 semanas? Pegue a música “Baby Don’t Cry” de 2 Pac, por exemplo. A saída acima sugere que ela esteve no top 100 por apenas 7 semanas, e todas as semanas restantes são preenchidas com valores ausentes (NA). Esses NAs na verdade não representam observações desconhecidas; eles foram forçados a existir pela estrutura do conjunto de dados. Altere o código usado em a) para remover esses NAs. Responda: Quantas linhas sobraram? (Dica: veja a documentação da função pivot_longer.)
c) Você deve ter percebido que no resultado do item a), o tipo da coluna week é caractere. Faça a transformação adequada para obter uma coluna com valores numéricos.
d) Qual música ficou por mais semanas no top 100 da Billboard em 2000? Por quantas semanas essa música apareceu no ranking? E qual música ficou por menos tempo no ranking.
e) Qual música ficou exatamente 10 semanas no top 100 da Billboard em 2000? Caso exista mais de uma música nessa condição, considere a que primeiro entrou no ranking.